Время прочтения: 4 мин.
Перед тем как рассказать о нашем исследовании, стоит отметить, что на данный момент Россия находится на 108 месте в топе стран с вакцинированным населением (53,3% является привитым по данным проекта Out World in Data на 11.12). Анализировать мы решили данные из репозитория университета Джонса Хопкинса на GitHub, но немного разобравшись в информации поняли, что он берёт их с сайта стопкоронавирус.рф.
Взглянем на исходную информацию. На рисунке 1 показан импорт и вид исходных данных.
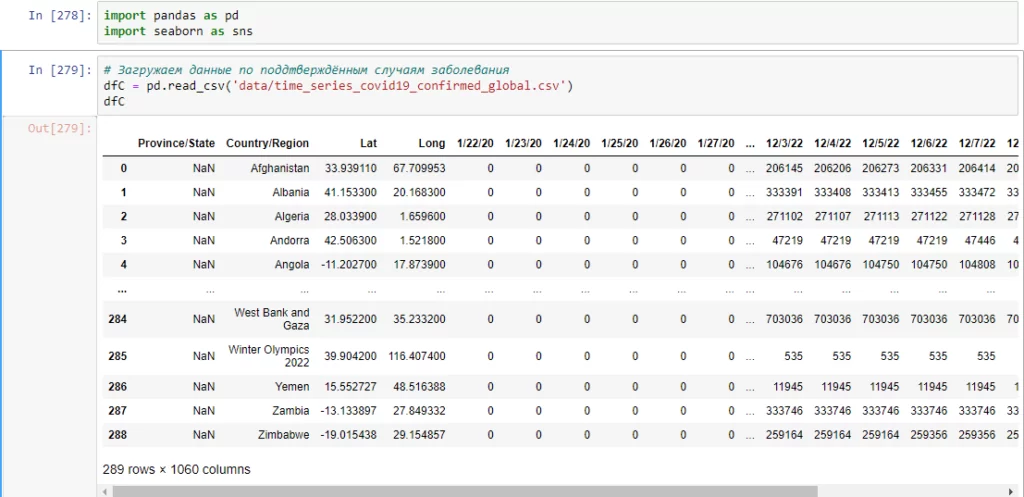
Итого перед нами оказалась картина о распространении коронавируса по 289 странам по дням, начиная с 22 января 2020 года по 12 декабря 2022. Среди некоторых стран известна также статистика распространения по регионам. Так же у нас есть аналогичные данные, касающиеся умерших и поправившихся (итого 3 датафрейма).
Уже на этом моменте можно заметить отсутствие множества данных, особенно в начальном периоде. Так что исходную информацию в любом случае необходимо подвергнуть предобработке.
Предобработка данных.
Так как нас больше заботит вопрос: “А что там в России?”, то мы отфильтровали получившийся датафрейм, исключив другие страны. Ниже показан код, с помощью которого мы это сделали.
Также мы удалили не интересующие нас столбцы, а именно: Province/State (данные для России о распространении по регионам отсутствуют), Country/Region (так как у нас будет одна страна) и другие. Для удобства дальнейшего анализа провели транспонирование датафрейма.
# Оставляем только Россию
dfC = dfC.loc[dfC['Country/Region'] == 'Russia']
# Убираем не интересующие нас столбцы
dfC = dfC.drop(['Province/State', 'Country/Region','Lat','Long'], axis = 1)
# Транспонируем датафрейм
dfCT = dfC.T
# Переименование столбца
dfCT = dfCT.rename(columns= {221:"Количество заболевших"})
dfCT
На рисунке 2 представлен результат выполнения этого кода.
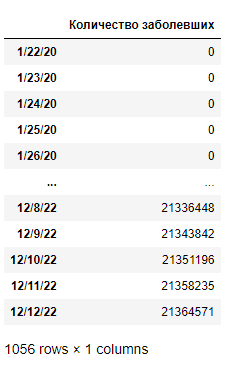
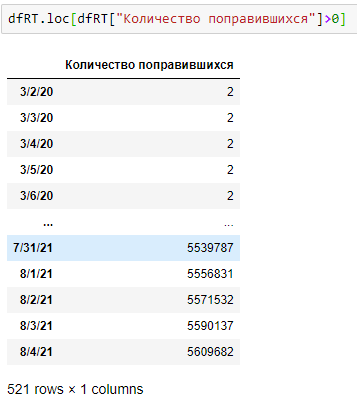
Исходя из рисунка 2, как было сказано ранее, можно заметить отсутствие данных за некоторый начальный период. Уберем его из рассмотрения. Реализующий код и его результат показан ниже (рисунок 3).
# отсекаем первые дни, где отсутствуют заболевшие
dfCT = dfCT.loc[dfCT['Количество заболевших']>0]
dfCT
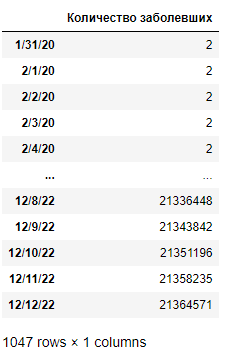
Из результата видно, что присутствуют только 2 заболевших с 31 января 2020 — единственные заболевшие до 2 марта 2020. Мы удалили данный период, ограничив его аналогичным образом, который может негативно повлиять на качество прогноза.
Далее от остальных 2-х датафреймов мы отсекли по 40 первых наблюдений (дней) для равенства выборок. Из количества умерших можно отбросить и больше, но это уже сместит данные о начальных смертях.
После этого собрали все транспонированные датафреймы в один с 3-мя столбцами, а именно Количество заболевших, Количество смертей и Количество поправившихся. В итоге у нас получился датафрейм из 3 столбцов и 1016 строк.
Визуализация данных.
Продолжая анализ, решили добавить визуализацию данных. Для этого использовали библиотеку seaborn (кто до сих пор о ней не слышал, рекомендую ознакомиться).
Однако после построения общего графика, показанного на рисунке 4, можно заметить в столбце с количеством поправившихся отсутствие множества данных, а именно из 1016 строк (количество дней), ненулевые значения содержатся только в 521 наблюдении и, начиная с 4 августа 2021 года, отсутствует. Учитывая высокую летальность при заболевании COVID-19 это похоже просто на отсутствующие данные. Конечно, можно было бы заполнить этот датафрейм какой-нибудь скользящей средней и т.д., но так как у нас есть другие данные, то отбросим их полностью.
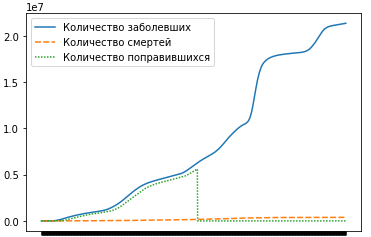
И на этом мы завершили нашу предобработку и визуализацию данных. Помните, что обработка исходной информации и грамотное её отсечение, порой являются задачей более важной, чем работа над самими данными. Ведь если в них присутствуют выбросы, это может испортить показания множества моделей.
Для построения прогноза мы используем датафрейм, состоящий из столбцов с количеством заболевших и количеством смертей, включающих в себя 1016 наблюдений вполне достаточных для построения прогнозирующей модели. Визуализация данных представлена на рисунке 6.
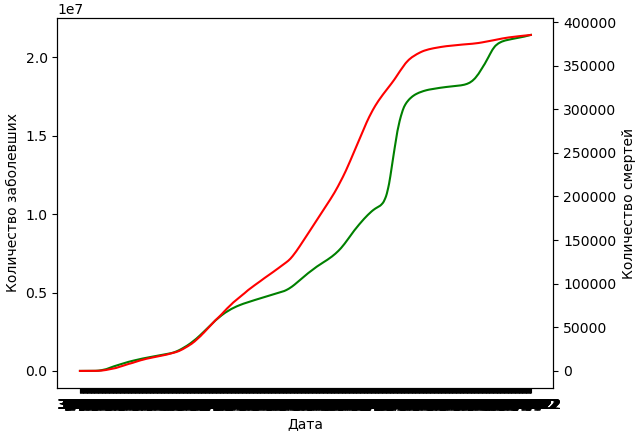
Полный код доступен по ссылке: https://github.com/MikhailOznobikhin/covid2022.

/img/star (2).png.webp)





/img/news (2).png.webp)
/img/event.png.webp)